Home > Structure > Department of Gene Expression > Gene Expression
Gene Expression
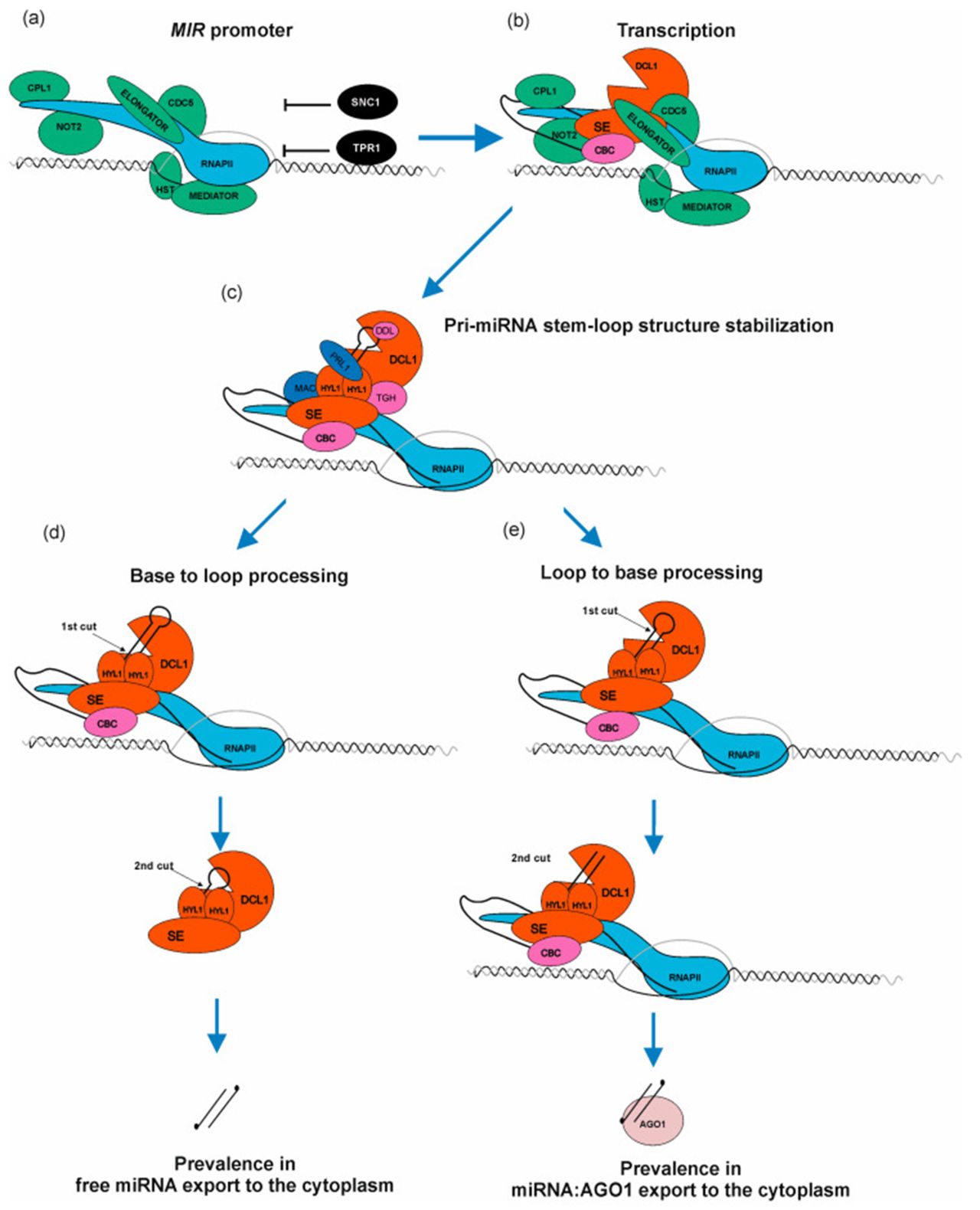
Our lab is working on gene expression regulation in plants. Specific emphasis is given to transcriptional and posttranscriptional regulation of microRNA biogenesis and microRNA function. Model plants as well as crop plants are studied.

Prof. Zofia Szweykowska-Kulińska
Principal Investigator

Prof. Artur Jarmołowski
Principal Investigator
Research
Our lab is working on gene expression regulation in plants. Specific emphasis is given to transcriptional and posttranscriptional regulation of microRNA biogenesis and microRNA function. Model plants like Arabidopsis thaliana, Marchantia polymorpha as well as crop plants like Hordeum vulgare and Solanum tuberosum are used. We focus on mechanistic aspects of microRNA biogenesis in model plants while function of microRNAs and the mode of their level control are studied in crop plants. Specifically we work on splicing, modification, polyadenylation and degradation of microRNA primary transcripts. The role microRNAs in plant development and response to environmental cues in crop plants are studied.
Selected publications
- SERRATE: a key factor in coordinated RNA processing in plants. Jozwiak M, Bielewicz D, Szweykowska-Kulinska Z, Jarmolowski A, Bajczyk M. Trends Plant Sci 2023.:S1360-1385(23)00089-4. doi: 10.1016/j.tplants.2023.03.009
- MicroRNA172b-5p/trehalose-6-phosphate synthase module stimulates trehalose synthesis and microRNA172b-3p/AP2-like module accelerates flowering in barley upon drought stress. (2023) Swida-Barteczka A, Pacak A, Kruszka K, Nuc P, Karlowski WM, Jarmolowski A, Szweykowska-Kulinska Z. Front Plant Sci 2023.;14:1124785. doi: 10.3389/fpls.2023.1124785.
- Chromatin-associated microprocessor assembly is regulated by the U1 snRNP auxiliary protein PRP40. Stepien A, Dolata J, Gulanicz T, Bielewicz D, Bajczyk M, Smolinski DJ, Szweykowska-Kulinska Z, Jarmolowski A. Plant Cell 2022.;34(12):4920-4935. doi: 10.1093/plcell/koac278.
- Biogenesis, conservation, and function of miRNA in liverworts. () Pietrykowska H, Sierocka I, Zielezinski A, Alisha A, Carrasco-Sanchez JC, Jarmolowski A, Karlowski WM, Szweykowska-Kulinska Z. J Exp Bot 2022.;73(13):4528-4545. doi: 10.1093/jxb/erac098.
- R-loops at microRNA encoding loci promote co-transcriptional processing of pri-miRNAs in plants. Gonzalo L, Tossolini I, Gulanicz T, Cambiagno DA, Kasprowicz-Maluski A, Smolinski DJ, Mammarella MF, Ariel FD, Marquardt S, Szweykowska-Kulinska Z, Jarmolowski A, Manavella PA. Nat Plants 2022.:402-418. doi: 10.1038/s41477-022-01125-x.
- mRNA adenosine methylase (MTA) deposits m6A on pri-miRNAs to modulate miRNA biogenesis in Arabidopsis thaliana. Bhat SS, Bielewicz D, Gulanicz T, Bodi Z, Yu X, Anderson SJ, Szewc L, Bajczyk M, Dolata J, Grzelak N, Smolinski DJ, Gregory BD, Fray RG, Jarmolowski A, Szweykowska-Kulinska Z. Proc Natl Acad Sci U S A. 2020; 117(35):21785-21795. doi: 10.1073/pnas.2003733117
- SERRATE interacts with the nuclear exosome targeting (NEXT) complex to degrade primary miRNA precursors in Arabidopsis. () Bajczyk M, Lange H, Bielewicz D, Szewc L, Bhat SS, Dolata J, Kuhn L, Szweykowska-Kulinska Z, Gagliardi D, Jarmolowski A. Nucleic Acids Res 2020.;48(12):6839-6854. doi: 10.1093/nar/gkaa373
- Active 5′ splice sites regulate the biogenesis efficiency of Arabidopsis microRNAs derived from intron-containing genes. Knop K, Stepien A, Barciszewska-Pacak M, Taube M, Bielewicz D, Michalak M, Borst JW, Jarmolowski A, Szweykowska-Kulinska Z. (2017) Nucleic Acids Res.; 45(5):2757-2775. doi: 10.1093/nar/gkw895
- Arabidopsis microRNA expression regulation in a wide range of abiotic stress responses. Barciszewska-Pacak M, Milanowska K, Knop K, Bielewicz D, Nuc P, Plewka P, Pacak AM, Vazquez F, Karlowski W, Jarmolowski A, Szweykowska-Kulinska Z. Front Plant Sci 2015.;6:410. doi: 10.3389/fpls.2015.00410.
- Down-regulation of CBP80 gene expression as a strategy to engineer a drought-tolerant potato. Pieczynski M, Marczewski W, Hennig J, Dolata J, Bielewicz D, Piontek P, Wyrzykowska A, Krusiewicz D, Strzelczyk-Zyta D, Konopka-Postupolska D, Krzeslowska M, Jarmolowski A, Szweykowska-Kulinska Z. Plant Biotechnol J 2013.;11(4):459-69. doi: 10.1111/pbi.12032.
More publications in PubMed.
Research grants
- Artur Jarmołowski:
2018/31/F/NZ2/03740 – BEETHOVEN LIFE 1, U1 snRNP i poliadenylacja – nowe powiązania, okres realizacji 27.01.2020 – 26.01.2026
2013/10/A/NZ1/00557 – MAESTRO 5, Molekularne interakcje pomiędzy białkami kompleksu dojrzewania mikroRNA i czynnikami odpowiedzialnymi za splicing i poliadenylację u roślin, okres realizacji 20.05.2014 – 19.05.2019 - Zofia Szweykowska-Kulińska
2020/39/B/NZ3/00539, OPUS 20, Sieć powiązań wątrobowcowo-specyficznych mikroRNA i ich docelowych mRNA w rozmnażaniu generatywnym Marchantia polymorpha (A network of liverwort-specific microRNAs and their targets in the sexual reproduction of Marchantia polymorpha), okres realizacji: 24.06.2021 – 23.06.2025
2012/04/M/NZ2/00127, HARMONIA 2, Regulacja ekspresji genów mikroRNA Arabidopsis thaliana w odpowiedzi na wybrane stresy abiotyczne: rola czynników transkrypcyjnych i splicingowych w biogenezie mikroRNA (Regulation of expression of Arabidopsis thaliana microRNA genes in response to selected abiotic stresses: the role of transcription and splicing factors in microRNA biogenesis.), okres realizacji 28.09.2012 – 27.09.2015
2016/23/B/NZ9/00862, OPUS 12, Sieci funkcjonalnych zależności pomiędzy mikroRNA z rodziny MIR444 a ich docelowymi czynnikami transkrypcyjnymi z rodziny MADS-box w rozwoju i odpowiedzi jęczmienia na stresy środowiskowe (Functional network of microRNA444 family members and their target MADS-box transcription factors in barley development and response to environmental stresses.), okres realizacji: 29.09.2017 – 28.09.2022 - Jakub Dolata
2017/25/B/NZ1/00603, OPUS 13 Nowa rola białka ARGONAUTE1 w biogenezie miRNA oraz splicingu pre-mRNA u Arabidopsis thaliana (A role for ARGONAUTE1 protein in miRNA biogenesis and pre-mRNA splicing in Arabidopsis thaliana), okres realizacji: 12.01.2018 – 11.01.2023 - Dawid Bielewicz
2022/45/B/NZ1/01273, OPUS 23, Konsekwencje modyfikacji potranslacyjnych białka HYL1 na rozwój roślin (Consequences of posttranslational modifications of HYL1 for plant development), okres realizacji: 10.01.2023 – 09.01.2027 - Mateusz Bajczyk
2021/41/B/NZ1/03644, OPUS 21, Udział helikaz typu DEAD-box w zależnej od SERRATE rekrutacji RBPs (białek wiążących RNA) do transkryptów polimerazy RNA II u roślin, okres realizacji: 12.01.2022 – 11.01.2026
Current group members

Prof. Zofia Szweykowska-Kulińska
principal investigator
more…

Prof. Artur Jarmołowski
principal investigator
more…

Andrzej Pacak, PhD, DSc
principal investigator
more…

Katarzyna Kruszka, PhD
senior specialist
more…

Halina Pietrykowska, PhD
senior specialist
more…

Aleksandra Świda-Barteczka, PhD
postdoc
more…

Izabela Sierocka, PhD
postdoc
more…

Dawid Bielewicz, PhD
postdoc
more…